MNIST 데이터셋으로 필기 인식 모델 만들기
이번 프로젝트도 오류가 많아서, 설명하면서 정정하려고 한다.
앞에서는 DeepChem에 사전 정의된 모델인 dc.models.MultitaskClassifier를 사용해 머신러닝을 했다. 그러나 사전 정의된 모델보다 사용자가 직접 모델을 정의하는 일이 더 많다. 직접 합성곱 신경망 모델을 만들어 MNIST 필기 인식 데이터셋에 적용시키는 방법을 배운다.
MNIST 필기 인식 데이터셋은 필기된 숫자를 올바르게 분류하는 머신러닝에 많이 사용된다. 전체적인 구조는 이미지의 일부 피처를 식별하기 위한 두 개의 합성곱 레이어로 시작해서 두 개의 완전 연결 레이어(full connection layer)를 통해 이미지에 적힌 숫자를 예측한다.
시작하기에 앞서 터미널에 다음 명령어를 입력해 MNIST 데이터를 다운로드한다.

파이썬으로 MNIST 데이터셋을 불러온다. 책에 적힌 코드는 다음과 같으나…
from tensorflow.examples.tutorials.mnist import input_data
mnist = input_data.read_data_sets("MNIST_data/", one_hot = True)
위 코드는 tensorflow 2.X.X 버전 이상일 경우 더 이상 제공하지 않는다는 것이었다.
따라서 아래 코드로 mnist 데이터를 불러올 수 있다.
mnist = tf.keras.datasets.mnist
필요한 라이브러리를 불러온다. 그런데 Tensorgraph 클래스는 이제 쓰이지 않는 코드이다.
import deepchem.models.tensorgraph.layers as layers
따라서 아래 (세 번째) 코드로 불러와야한다.
import deepchem as dc
import tensorflow as tf
from deepchem.models import GraphConvModel
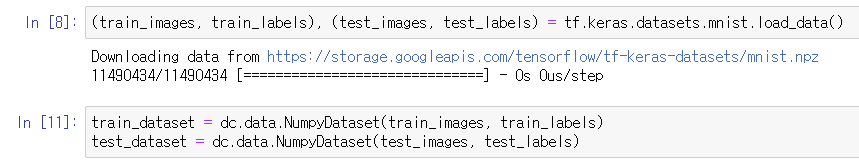
MNIST 데이터셋을 학습 데이터와 테스트 데이터로 변환한다.
신경망의 각 레이어는 이전 레이어의 입력을 받아 이후 레이어로 전달하는 출력을 계싼한다. 신경망의 가장 앞에는 데이터셋의 피처 및 레이블 값을 받는 입력 레이어가 있고, 가장 끝에는 수행한 계산의 결과를 반환하는 출력 레이어가 있다. 이미지 처리용 합성곱 신경망을 만들기 위해 모델을 정의한다.
model = GraphConvModel(model_dir='mnist', n_tasks = 2)
여기서 model_dir 옵션은 모델의 매개변수를 저장하는 폴더를 지정하기 위한 것이다. 해당 옵션을 생략하면 모델이 저장되지 않기 때문에 파이썬 인터프리터가 종료되면 모든 작업 내용이 사라진다. 지정 폴더를 지정하면 이후 필요할 때 해당 모델을 다시 불러올 수 있다.
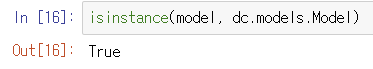
GraphConvModel은 Model 클래스를 상속하므로 dc.models.Model.dml 인스턴스(instance)이다.
여기까지는 어렵지 않게 했으나, 이하부터는 대체 코드를 찾기 어려워서 일단 책 내용을 정리하려고 한다.
https://forum.deepchem.io/t/deepchem-2-3-keras-modeling-and-groundwork-for-tensorflow-2/61
DeepChem 2.3: Keras modeling and groundwork for TensorFlow 2
DeepChem 2.3 includes many architectural changes. Some are mostly invisible, while others will directly affect how you work with it. All of them have the same goal: to make sure DeepChem can continue to function well as part of the larger TensorFlow ecosys
forum.deepchem.io
위 홈페이지에서 TensorGraph Version을 Keras Version으로 바꾼 코드를 알려줬으나, 모델을 학습시키는 fit() 구간부터 코드를 어떻게 건드려야 할지 몰라 이하 내용은 책 내용 그대로 서술하겠다.
Feature 및 Label 클래스를 추가해 피처와 레이블에 대한 입력을 받을 수 있게 한다.
feature = layers.Feature(shape=(None, 784))
label = layers.Label(shape=(None, 10))MNIST 데이터셋은 28 × 28 픽셀의 이미지 파일로 구성돼 있다. 각각의 이미지는 피처가 784개인 벡터로 변환해 입력 데이터로 사용되고, 이미지의 레이블은 0부터 9까지 총 열 개가 있다. 텐서플로에서 기본적으로 None을 옵션 값으로 사용하면 모든 크기를 허용한다는 의미가 된다. 위의 코드에서 shape 옵션의 첫 번째 요소는 배치의 크기를 의미하기 때문에 우리가 만든 모델은 배치 크기와 무관하게 작동한다.
합성곱 레이어를 적용하려면 현재 28 × 28 형태의 피처 값은 Reshape() 레이어를 써서 평평한 1차원 데이터로 만들어준다.
make_image = layers.Reshape(shape=(None, 28, 28), in_layers = feature)위에서 옵션에 사용한 None 값은 임의의 배치 크기를 처리할 수 있다는 의미다. 키워드 인수 in_layers = feature는 Reshape 레이어가 이전 Feature 레이어의 피처를 입력으로 사용한다는 것을 나타낸다.
conv2d_1 = layers.Conv2D(
num_outputs=32, activation_fn=tf.nn.relu, in_layers=make_image)
conv2d_2 = layers.Conv2D(
num_outputs=64, activation_fn=tf.nn.relu, in_layers=conv2d_1)
in_layer 옵션을 사용해 레이어들을 연결한다. Conv2D 레이어의 출력은 2차원 벡터이므로 최종 출력 레이어를 연결하려면 평평하게 1차원 벡터로 변환해야 한다. 정확히 말하면, Conv2D 레어는 각 샘플에 대해 2차원 출력값을 생성하므로 출력값에는 샘플에 대한 정보도 포함돼 3차원 벡터가 된다. Flatten 레이어는 이것을 샘플당 하나의 차원으로 총 2차원 벡터로 축소한다.
flatten = layers.Flaten(in_layers=conv2d_2)
dense1 = layers.Dense(
out_channels=1024, activation_fn=tf.nn.relu, in_layers=flatten)
dense2 = layers.Dense(
out_channels=10, activation_fn=None, in_layers=dense1)
정확한 머신러닝을 위해 두 번째 레이어를 손실 함수로 연결해야 한다. 손실 함수 SoftMaxCrossEntropy를 사용한다.
smce = layers.SoftMaxCrossEntropy(in_layers=[label, dense2])
loss = layers.ReduceMean(in_layers=smce)
model.set_loss(loss)모든 샘플에 대한 손실 함수의 값을 계산하기 위해 최종 손실 값은 모든 샘플에 대한 평균으로 구한다.
소프트맥스 교차 엔트로피(SoftMaxCrossEntropy)는 먼저 소프트맥스 함수를 사용해 출력값을 확률로 변환한 다음, 레이블로 해당 확률의 교차 엔트로피를 계산한다. 레이블은 원-핫 인코딩된 것이다. 즉, 올바르게 분류한 경우에는 1, 다른 잘못된 분류인 경우에는 0이다. 정확한 분류로 예측된 확률이 1에 가까울수록 손실은 최소화된다. 이 두 가지 연산은 종종 동시에 나타나며 이를 단일 단계로 계산하면 개별적으로 수행하는 것보다 안정적인 수치를 얻을 수 있다.
# 각각의 분류에 따른 확률을 얻기 위해 SoftMax 레이어로 출력을 변환한다.
output = layers.SoftMax(in_layers=dense2)
# 출력을 모델에 추가한다.
model.add_output(output)
# fit() 함수를 사용해 모델을 학습시킨다.
model.fit(train_dataset, nb_epoch=10)
# 정확도를 계산한다.
metric = dc.metrics.Metric(dc.metric.accuracy_score)
train_scores = model.evaluate(train_dataset, [metric])
test_scores = model.evaluate(test_dataset, [metric])
'AI / DL > 생명과학을 위한 딥러닝' 카테고리의 다른 글
| [생명과학을 위한 딥러닝] 4장. 분자 수준 데이터 다루기 (0) | 2022.10.18 |
|---|---|
| [생명과학을 위한 딥러닝] 3장. DeepChem을 이용한 머신러닝 (1) (0) | 2022.10.15 |
| [생명과학을 위한 딥러닝] 2장. 딥러닝 소개 (0) | 2022.10.03 |



댓글